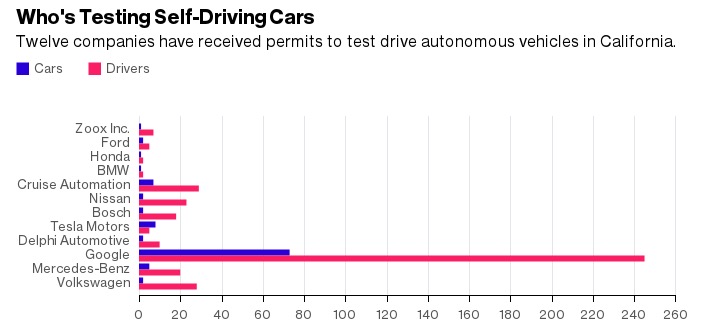
gganimate|让你的图动起来!!!
这是ggplot中十分可爱的一个扩增包,目的只有一个,就是让你的图动起来!就是酱紫!!
gganimate扩展了ggplot2实现的图形语法,包括动画描述。它通过提供一系列新的语法类来实现这一点,这些类可以添加到绘图对象中,以便自定义它应该如何随时间变化。
下面是他的parameter:
transition_*()定义了数据应该如何展开以及它与时间的关系。view_*()定义位置比例应如何沿动画更改。shadow_*()定义如何在给定的时间点呈现来自其他时间点的数据。enter_*()/ exit_*()定义新数据应如何显示以及旧数据在动画过程中应如何消失。ease_aes()定义了在过渡期间应该如何进行过渡。
举个栗子!
#安装辅助包,该包有两个版本,已经更新为最新版本,老版本在未来将不再支持。install.packages("gganimate")# 安装开发版
# install.packages('devtools')
# devtools::install_github('thomasp85/gganimate')
library(ggplot2)
library(gganimate)ggplot(mtcars, aes(factor(cyl), mpg)) +geom_boxplot() + geom_point() +# Here comes the gganimate codetransition_states(gear,transition_length = 2,state_length = 1) +enter_fade() +exit_shrink() +ease_aes('sine-in-out')加载时间是比较长的,需要耐心等待哈!
Yet Another Example
首先查看一下数据格式吧,Gapminder是关于预期寿命,人均国内生产总值和国家人口的数据摘录。
library(gapminder)
head(gapminder)#我们看一下数据格式ggplot(gapminder, aes(gdpPercap, lifeExp, size = pop, colour = country)) +
#点的大小和颜色分别由pop和country决定;geom_point(alpha = 0.7, show.legend = FALSE) +scale_colour_manual(values = country_colors) + #进行数值之间的映射scale_size(range = c(2, 12)) + #设置绘图符号大小scale_x_log10() + #连续数据位置的标准化facet_wrap(~continent) + #按照continent进行分类# Here comes the gganimate specific bitslabs(title = 'Year: {frame_time}', x = 'GDP per capita', y = 'life expectancy') +transition_time(year) +ease_aes('linear')#指数据变化的状态,线性发展比较缓慢
哈哈哈,现在我们以肿瘤数据为例进行演示一下:
我编了一组测试数据,其中将肿瘤分为I,II,III型,IV型为control,然后分别显示了再不同样本中不同肿瘤分型下的部分基因的表达情况。
library(ggplot2)
library(gganimate)
#首先我们进行数据的读入data <- "subgroup,sample,gene,expression
I,Tumor,p53,12.725952
II,Tumor,p53,11.914176
III,Tumor,p53,12.315768
IV,Normal,p53,12.978894
I,Tumor,p53,11.93924
II,Tumor,p53,12.262185
III,Tumor,p53,11.538924
IV,Normal,p53,12.016589
I,Tumor,p53,12.302574
II,Tumor,p53,11.939233
III,Tumor,p53,12.803992
IV,Normal,p53,10.674506
I,Tumor,p53,12.569142
II,Tumor,p53,12.088496
III,Tumor,p53,9.971951
IV,Normal,p53,13.008554
I,Tumor,p53,12.804154
II,Tumor,p53,11.847107
III,Tumor,p53,12.081261
IV,Normal,p53,12.158431
I,Tumor,p53,11.096693
II,Tumor,p53,12.655811
III,Tumor,p53,11.509067
IV,Normal,p53,12.523573
I,Tumor,p53,11.3554
II,Tumor,p53,11.560566
III,Tumor,p53,10.969046
IV,Normal,p53,11.169892
I,Tumor,p53,12.884054
II,Tumor,p53,12.284268
III,Tumor,her2,9.575523
IV,Normal,her2,12.409381
I,Tumor,her2,12.114364
II,Tumor,her2,11.493997
III,Tumor,her2,10.987106
IV,Normal,her2,11.943991
I,Tumor,her2,11.171378
II,Tumor,her2,13.120248
III,Tumor,her2,12.628872
IV,Normal,her2,11.91914
I,Tumor,her2,12.36504
II,Tumor,her2,12.707354
III,Tumor,her2,12.54517
IV,Normal,her2,12.199749
I,Tumor,her2,13.184496
II,Tumor,her2,12.640412
III,Tumor,her2,12.716897
IV,Normal,her2,13.359091
I,Tumor,her2,11.760945
II,Tumor,her2,11.406367
III,Tumor,her2,11.984382
IV,Normal,her2,12.254977
I,Tumor,her2,11.579763
II,Tumor,her2,11.983042
III,Tumor,her2,12.566317
IV,Normal,her2,10.869331
I,Tumor,her2,10.910963
II,Tumor,her2,11.948207
III,Tumor,myc,12.363072
IV,Normal,myc,12.755182
I,Tumor,myc,11.922223
II,Tumor,myc,9.618839
III,Tumor,myc,12.693868
IV,Normal,myc,13.40685
I,Tumor,myc,11.871609
II,Tumor,myc,11.783704
III,Tumor,myc,12.485053
IV,Normal,myc,12.669123
I,Tumor,myc,11.653691
II,Tumor,myc,11.675768
III,Tumor,myc,12.744605
IV,Normal,myc,12.911619
I,Tumor,myc,12.008307
II,Tumor,myc,11.838161
III,Tumor,myc,12.590989
IV,Normal,myc,11.680278
I,Tumor,myc,11.719241
II,Tumor,myc,10.156746
III,Tumor,myc,11.84406
IV,Normal,myc,12.975393
I,Tumor,myc,10.963332
II,Tumor,myc,12.338216
III,Tumor,myc,12.030859
IV,Normal,myc,11.119114
I,Tumor,myc,12.661349
II,Tumor,myc,13.168166
III,Tumor,myc,11.707595
IV,Normal,myc,12.06719
I,Tumor,myc,12.463962
II,Tumor,myc,12.288819
III,Tumor,myc,12.036757
IV,Normal,myc,12.98055
I,Tumor,myc,11.343075
II,Tumor,myc,12.565481
III,Tumor,myc,12.279996
IV,Normal,myc,12.965189
I,Tumor,myc,12.946155
II,Tumor,myc,11.688462
III,Tumor,sox4,11.944477
IV,Normal,sox4,12.128177
I,Tumor,sox4,11.116105
II,Tumor,sox4,11.148871
III,Tumor,sox4,13.139244
IV,Normal,sox4,10.043207
I,Tumor,sox4,12.043914
II,Tumor,sox4,9.990576
III,Tumor,sox4,11.624263
IV,Normal,sox4,11.647402
I,Tumor,sox4,12.502176
II,Tumor,sox4,12.291812
III,Tumor,sox4,11.421913
IV,Normal,sox4,12.282511
I,Tumor,sox4,12.511991
II,Tumor,sox4,12.285322
III,Tumor,sox4,11.7884
IV,Normal,sox4,13.747552
I,Tumor,sox4,11.212993
II,Tumor,sox4,12.936845
III,Tumor,sox4,12.442484
IV,Normal,sox4,10.324288
I,Tumor,sox4,12.436421
II,Tumor,sox4,11.923122
III,Tumor,sox4,12.831474
IV,Normal,sox4,12.271537
I,Tumor,sox4,12.208086
II,Tumor,sox4,11.830799
III,Tumor,sox4,12.410238
IV,Normal,sox4,12.13912
I,Tumor,sox4,12.47"test <- read.csv(text=data,header=T)
head(test)library(ggplot2)
ggplot(test,aes(x=subgroup,y=expression,fill=subgroup))+geom_boxplot()+geom_jitter()+theme_bw() #按照subgroup进行分型,并画出箱式图同样对不同gene在各组中的分布情况进行描述:
library(ggplot2)
p <- ggplot(test,aes(x=subgroup,y=expression,fill=subgroup))+geom_boxplot()+geom_jitter()+theme_bw()
p +facet_grid(.~gene)#按照gene对各个小组进行分类library(ggplot2)
library(gganimate)
p <- ggplot(test,aes(x=subgroup,y=expression,fill=subgroup))+geom_boxplot()+geom_jitter()+theme_bw()
p +transition_states(gene, state_length = 0)+labs(title = "{closest_state} expression")
想不想让你的数据动来动去,哈哈哈,不妨试试这个R包!
如果想学习更多有关该R包的情况,可以点击:https://github.com/thomasp85/gganimate。|||||作者:清华大学医学院 苑晓梅
猜你喜欢
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature Cell专刊 肠道指挥大脑
系列教程:微生物组入门 Biostar 微生物组 宏基因组
专业技能:学术图表 高分文章 生信宝典 不可或缺的人
一文读懂:宏基因组 寄生虫益处 进化树
必备技能:提问 搜索 Endnote
文献阅读 热心肠 SemanticScholar Geenmedical
扩增子分析:图表解读 分析流程 统计绘图
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
在线工具:16S预测培养基 生信绘图
科研经验:云笔记 云协作 公众号
编程模板: Shell R Perl
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外5000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。PI请明示身份,另有海内外微生物相关PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
点击阅读原文,跳转最新文章目录阅读